Analysesystem für Ultraschallsequenzen
Herz-Kreislauf-Erkrankungen sind die führende Todesursache in Deutschland und verursachen insgesamt etwa 40 Prozent aller Sterbefälle. Darüber hinaus sind sie mit erheblichen individuellen Krankheitsfolgen verbunden und verursachen hohe gesellschaftliche Krankheitskosten [1].
Durch eine rechtzeitige Ermittlung von Risikofaktoren könnten diese Erkrankungen aber frühzeitig erkannt und adäquate Therapiemaßnahmen eingeleitet werden. Gesundheitsdaten wie das Gewicht oder der Blutdruck von Patient:innen sind aber aber nur vergleichsweise schwache Indikatoren.
Die Wissenschaftler:innen an der Universität Basel untersuchen hingegen die Möglichkeit der Analyse von Ultraschallsequenzen der Halsschlagader. Die Aufnahme dieser Daten stellt eine niederschwellige Analysemethode dar, die keine aufwendigen Verfahren oder gar operative Eingriffe benötigt, sondern auch regelmäßig beim ansässigen Hausarzt durchgeführt werden könnte.
Bei der Analyse werden die Halsschlagader selbst, die Dicke der Gefäßwände (besonders die „Intima-Media-Dicke“) und mögliche Ablagerungen an jenen Gefäßwänden untersucht.
Die manuelle Auswertung der Ultraschallsequenzen für die Forschung erfordert dabei einen enormen Arbeitsaufwand. Für aktuelle Studien müssen oft tausende von Ultraschallsequenzen durchsucht und sortiert werden, um nur die Bilder zu erhalten, bei denen die Gefäßwände in der richtigen Lage deutlich sichtbar sind. Erst im Anschluss können die eigentlichen Messungen durchgeführt werden.
Unser Analysetool IMTsys erleichtert diese Arbeit, indem es die Wissenschaftler:innen bei der Vorauswahl unterstützt.
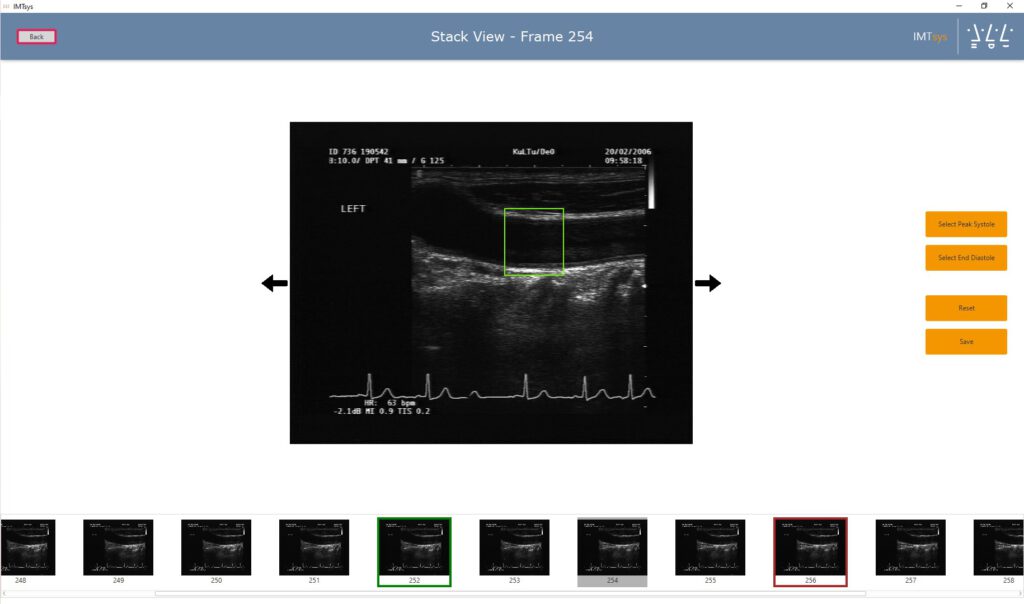
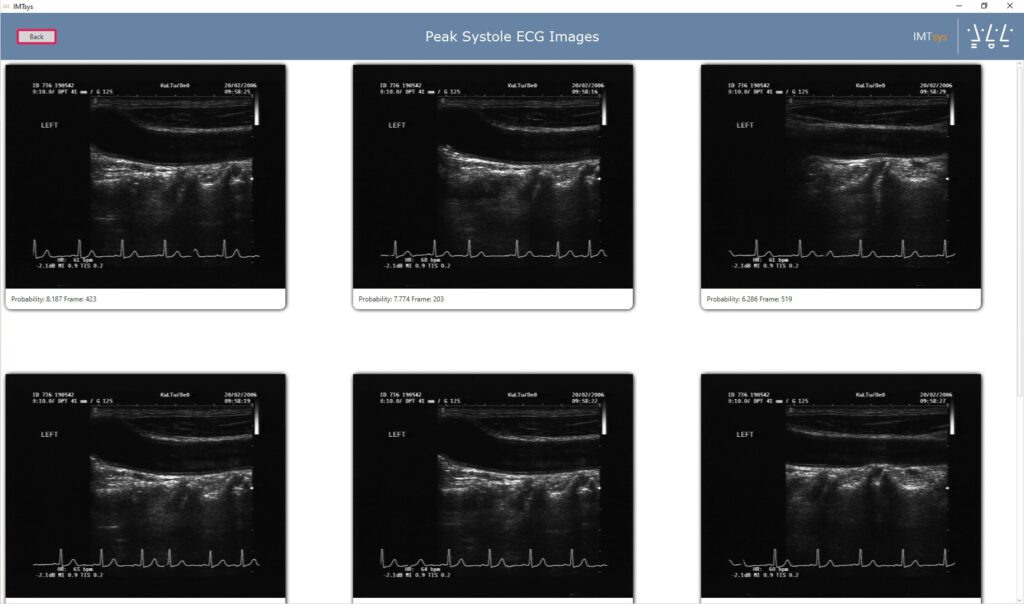
Mittels Bildverarbeitungsalgorithmen wird zunächst das EKG analysiert, um die für die Analyse optimalen Zeitpunkte im Herzrhythmus zu finden. Ein Optical-Flow-Algorithmus filtert anschließend unruhige Bilder. Schließlich wird durch Einsatz eines selbsttrainierten Neuronalen Netzes eine Bildsegmentierung durchgeführt.
So können durch die Vorverarbeitung zunächst alle unbrauchbaren Bilder gefiltert werden. Mithilfe des Neuronalen Netzes kann anschließend eine „Region of Interest“ bestimmt werden, innerhalb der die Gefäßwände gut sichtbar sind. Diese kann nun exportiert und anschließend direkt in das Tool zur Ultraschallmessung importiert werden, um die Messung durchzuführen. Dadurch wird der Zeitaufwand für die Forschenden deutlich reduziert.
Rahmendaten:
- Jahr: 2021
- Studierende: Anton Hummel, Que Linh Phan, Dorian Kemgang Mbeugang, Simon Müller, Fabien Zwick
- Semester: Master
- Studiengang: Informatik
- Supervision: Prof. Dr.-Ing. Alexandra Teynor, Prof. Dr. Arno Schmidt-Trucksäss
- Tags: IMT, Computer Vision, Python, JavaFx, FastAPI, Tensorflow, Deep Learning, U-net, Image Recognition

